Exemples d'entrées et de sorties de l'ensemble de données MADRC. Crédit: (2023). DOI : 10.7554/eLife.91398.1
Les chercheurs ont développé une suite d'outils gratuits pour analyser de grandes quantités de photographies de dissection cérébrale dans des banques de cerveaux du monde entier afin d'améliorer la compréhension des maladies neurodégénératives.
L'étude, publiée aujourd'hui sous forme de prépublication révisée dans eLifeest décrit par les éditeurs comme fournissant un outil open source précieux pour les chercheurs dans le domaine de la neuropathologie et de la neuroimagerie, étayé par des preuves convaincantes issues d'expériences utilisant à la fois des données réelles et synthétiques.
La mesure du volume de différentes régions du cerveau est un moyen important de comprendre le vieillissement et les maladies neurodégénératives et est généralement réalisée soit en utilisant une imagerie par résonance magnétique (IRM) chez des personnes de leur vivant, soit en étudiant des sections de tissus cérébraux données à des banques de cerveaux. après la mort.
Être capable de relier les résultats de l'analyse microscopique des tissus aux données macroscopiques à grande échelle provenant des examens IRM est inestimable, mais les examens IRM sont généralement obtenus sur des patients plusieurs années avant leur décès, ce qui rend difficile le lien entre ce qui est vu sur l'examen et ce qui est observé. vu plus tard au microscope.
Une alternative à cela consiste à effectuer une IRM du cerveau après l'autopsie et avant que les coupes de tissus ne soient prélevées pour une analyse microscopique. Cependant, peu de centres de biobanques disposent de l’équipement ou de l’expertise pour le faire. Au lieu de cela, les mesures quantitatives telles que l’épaisseur corticale et l’atrophie de régions spécifiques sont souvent estimées qualitativement par les chercheurs examinant des tranches de cerveau.
“Nous avons décidé de proposer une solution à ce problème en exploitant les photographies de dissection de tranches de cerveau acquises en routine avant l'analyse microscopique”, explique l'auteur principal Harshvardhan Gazula, associé de recherche postdoctoral au Centre Athinoula A. Martinos d'imagerie biomédicale du Massachusetts General Hospital (MGH). ), Massachusetts, États-Unis.
“Ces vastes collections de photographies de dissection présentent une ressource d'information inestimable et actuellement sous-utilisée, qui promet de faire progresser notre compréhension de diverses fonctions et troubles cérébraux.”
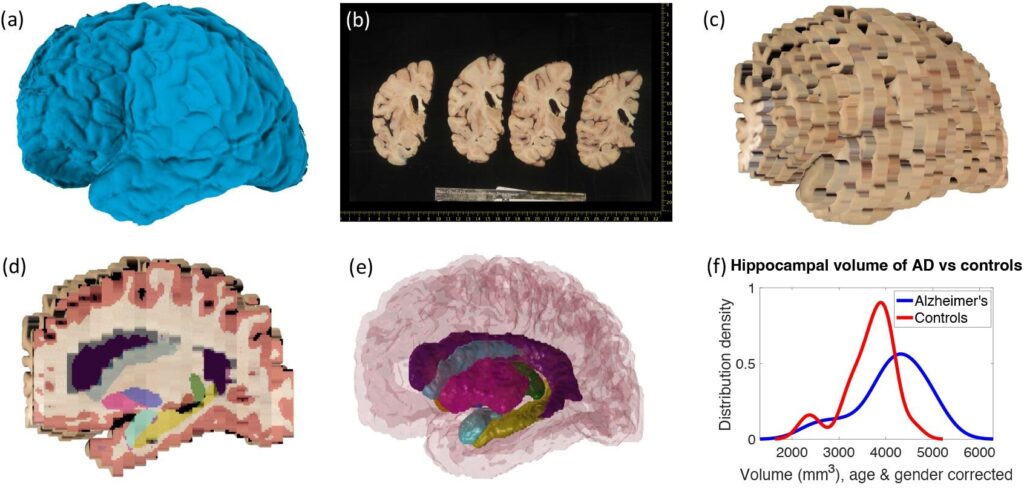
Harshvardhan et ses collègues ont développé une suite d'outils informatiques qui permettront à d'autres chercheurs d'utiliser ces photographies de dissection pour reconstruire une image 3D du cerveau. La suite est disponible gratuitement et comprend trois modules : le premier corrige les différentes perspectives et tailles de pixels dans les images originales ; la seconde construit une reconstruction 3D du cerveau à partir des photos, en utilisant une analyse 3D de la surface du cerveau ou un atlas cérébral générique comme point de référence ; et le troisième module segmente ensuite la reconstruction cérébrale en différentes régions cérébrales, telles que l'hippocampe, le thalamus et le cortex.
La structure et les volumes de ces régions peuvent ensuite être comparés à d’autres IRM et utilisés parallèlement aux données microscopiques pour en savoir plus sur les changements qui se produisent dans les maladies neurodégénératives.
Pour évaluer l’exactitude des outils, l’équipe a effectué trois étapes de validation. Tout d’abord, ils ont utilisé leur outil pour analyser des photographies de dissection de 21 cas confirmés post-mortem de la maladie d’Alzheimer et de 12 cerveaux témoins du même âge. Cela a montré que le modèle capturait des schémas bien connus de la maladie d'Alzheimer, tels que le rétrécissement (atrophie) de l'hippocampe et l'élargissement du ventricule cérébral.
Ensuite, ils ont comparé la précision du nouvel outil avec la méthode d’analyse de référence actuelle, qui utilise une technique différente pour l’étape de segmentation. Cela a montré que le nouvel outil surpassait l’ancienne version à plusieurs égards : par exemple, il était plus efficace pour combler les informations manquantes entre les tranches de cerveau.
Enfin, l’équipe a évalué la robustesse des algorithmes pour reconstruire le cerveau sur un ensemble important et variable de données simulées à partir d’IRM provenant de 500 autres participants. Cela a montré que la méthode était raisonnablement robuste même lorsqu’il y avait plus d’espacement entre les images de tranches de cerveau, mais cela a également montré que la cohérence de l’épaisseur des tranches de cerveau est importante pour la précision.
Cette étude présente quelques limites, notamment le fait que la méthode ne peut segmenter que l'ensemble du cortex et est actuellement incapable de le subdiviser en régions, une tâche connue sous le nom de « parcellation corticale ».
Une parcellisation corticale précise serait intéressante car la morphométrie des régions individuelles, en particulier les mesures de leur épaisseur, est plus prédictive de la neuropathologie. Les auteurs disent qu’ils cherchent maintenant à étendre l’ensemble d’outils pour permettre la parcellation corticale à l’avenir.
« Exploiter les vastes quantités de photographies de dissection disponibles dans les banques de cerveaux du monde entier pour effectuer la morphométrie est une voie prometteuse pour améliorer notre compréhension de diverses maladies neurodégénératives », conclut l'auteur principal Juan Eugenio Iglesias, professeur agrégé de radiologie au Centre Athinoula A. Martinos pour les études biomédicales. Imagerie à MGH.
« Nos outils accessibles au public sont faciles à utiliser par toute personne ayant peu ou pas de formation et permettent d'établir un lien rentable et rapide entre les phénotypes morphométriques et le diagnostic neuropathologique. Nous espérons que ces outils joueront un rôle crucial dans la découverte de nouveaux marqueurs d'imagerie. pour étudier les maladies neurodégénératives.
Plus d'information:
Harshvardhan Gazula et al, Apprentissage automatique de photographies de dissection et de numérisation de surface pour la neuropathologie 3D quantitative, eLife (2023). DOI : 10.7554/eLife.91398.1
eLife
Citation: De nouveaux outils informatiques peuvent reconstruire le cerveau en 3D à partir de photos de la biobanque (12 décembre 2023) récupéré le 12 décembre 2023 sur
Ce document est soumis au droit d'auteur. En dehors de toute utilisation équitable à des fins d'étude ou de recherche privée, aucune partie ne peut être reproduite sans autorisation écrite. Le contenu est fourni seulement pour information.