Crédit: Rapports de cellules (2023). DOI : 10.1016/j.celrep.2023.113355
Les chercheurs ont identifié une nouvelle façon de dépister les gènes responsables de la croissance de plusieurs types de cancers, identifiant des cibles particulièrement prometteuses pour l’oncologie de précision dans les cancers épidermoïdes de la bouche et de l’œsophage.
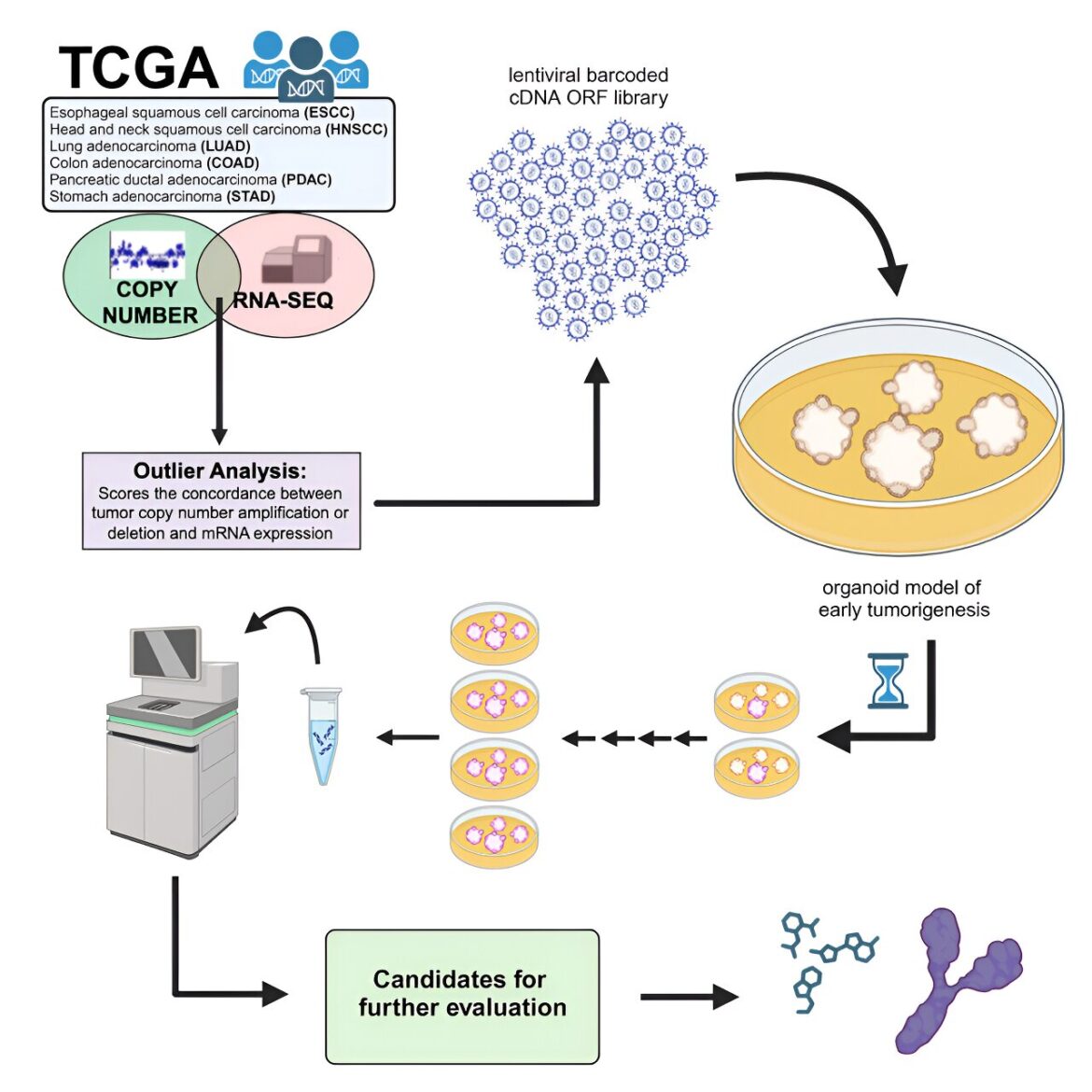
L’étude, publiée dans le numéro de ce mois-ci de Rapports de cellulesa utilisé des modèles tridimensionnels de tissus organiques appelés organoïdes pour identifier et tester des cibles génétiques potentielles de l’Atlas du génome du cancer.
“L’Atlas du génome du cancer contient une quantité énorme de données, et ce domaine a permis de développer des médicaments de précision qui prolongent et sauvent des vies. Mais seule une minorité de ces données nous indiquent comment les cancers se développent et s’il s’agit d’une cible médicamenteuse”, a expliqué l’auteur principal, le Dr. Ameen Salahudeen, professeur adjoint de médecine et de biochimie à l’Université de l’Illinois à Chicago, et ancien chercheur postdoctoral dans le laboratoire de Calvin Kuo à l’Université de Stanford lorsque la recherche a été lancée. “Nous avions besoin d’une méthode évolutive et fonctionnelle pour analyser les données en termes de facteurs déterminants de la croissance du cancer et de la possibilité de les cibler.”
Afin d’identifier les gènes responsables de la croissance des tumeurs, les chercheurs ont décidé de se concentrer sur les zones du génome qui présentaient deux éléments : des régions possédant des copies anormalement élevées du même gène, quelque chose de commun à de nombreux types de cancer, et des régions avec des niveaux élevés. de l’expression de l’ARN, ce qui indiquerait que ces gènes sont impliqués dans la croissance tumorale. Pour ce faire, ils ont utilisé un nouvel algorithme développé à Stanford par le Dr Jose Seoane, qui travaillait alors dans le laboratoire de Christina Curtis.
L’équipe a identifié des régions prometteuses dans le génome de six cancers différents : de l’œsophage, de la cavité buccale, du côlon, de l’estomac, du pancréas et du poumon. Ensuite, ils ont construit des organoïdes tumoraux (de petits tissus tumoraux dans une assiette) pour chacun des six organes et ont testé leurs gènes candidats sur les organoïdes pour voir lesquels étaient associés à la croissance des tumeurs.
L’utilisation d’organoïdes pour cette étape constitue une amélioration par rapport aux méthodes standard précédentes, a expliqué Salahudeen. Les lignées cellulaires immortalisées généralement utilisées dans la recherche sur le cancer cellulaire présentent souvent de nombreuses mutations supplémentaires survenant après des années de culture en laboratoire, “ce qui confond vraiment les choses”, a-t-il déclaré. Et tester autant de gènes potentiels chez la souris serait impossible à mettre à l’échelle et prendrait des années.
En collaboration avec le Dr David Root du Broad Institute et le Dr Bill Hahn du Dana Farber Cancer Institute, l’équipe a ensuite examiné les organoïdes à l’aide de bibliothèques de lentivirus de ces gènes. Les organoïdes ont révélé des résultats particulièrement intéressants pour les cancers épidermoïdes de la cavité buccale et de l’œsophage, qui possèdent tous deux très peu de gènes conducteurs connus qui pourraient être ciblés par des médicaments. En outre, les chercheurs ont testé une petite molécule disponible en clinique – un type de médicament connu sous le nom d’inhibiteur du FGFR – sur les organoïdes de l’œsophage et ont pu réduire la tumeur.
“Ce gène, FGF3, est amplifié dans près de la moitié des cancers épidermoïdes de l’œsophage et est connu pour interagir avec le FGFR”, a déclaré Salahudeen. “Cela pourrait donc potentiellement bénéficier à près de la moitié des patients atteints d’un cancer de l’œsophage.”
Salahudeen, qui est membre du Centre de lutte contre le cancer de l’Université de l’Illinois, a déclaré que la prochaine étape consiste à étudier les inhibiteurs cliniques existants du FGFR dans cette nouvelle indication chez les patients atteints d’un cancer épidermoïde de l’œsophage et à continuer d’étudier d’autres gènes potentiellement prometteurs identifiés par l’étude.
Parmi les autres auteurs de l’article figurent le co-auteur principal Kanako Yuki du laboratoire Calvin Kuo à Stanford.
Plus d’information:
Ameen A. Salahudeen et al, Criblage fonctionnel des oncogènes aberrants d’amplification dans les modèles organoïdes de tumorigenèse précoce, Rapports de cellules (2023). DOI : 10.1016/j.celrep.2023.113355
Fourni par l’Université de l’Illinois à Chicago
Citation: La culture de microtumeurs dans une boîte permet d’identifier rapidement les gènes qui pilotent la croissance tumorale (28 novembre 2023) récupéré le 28 novembre 2023 sur
Ce document est soumis au droit d’auteur. En dehors de toute utilisation équitable à des fins d’étude ou de recherche privée, aucune partie ne peut être reproduite sans autorisation écrite. Le contenu est fourni seulement pour information.