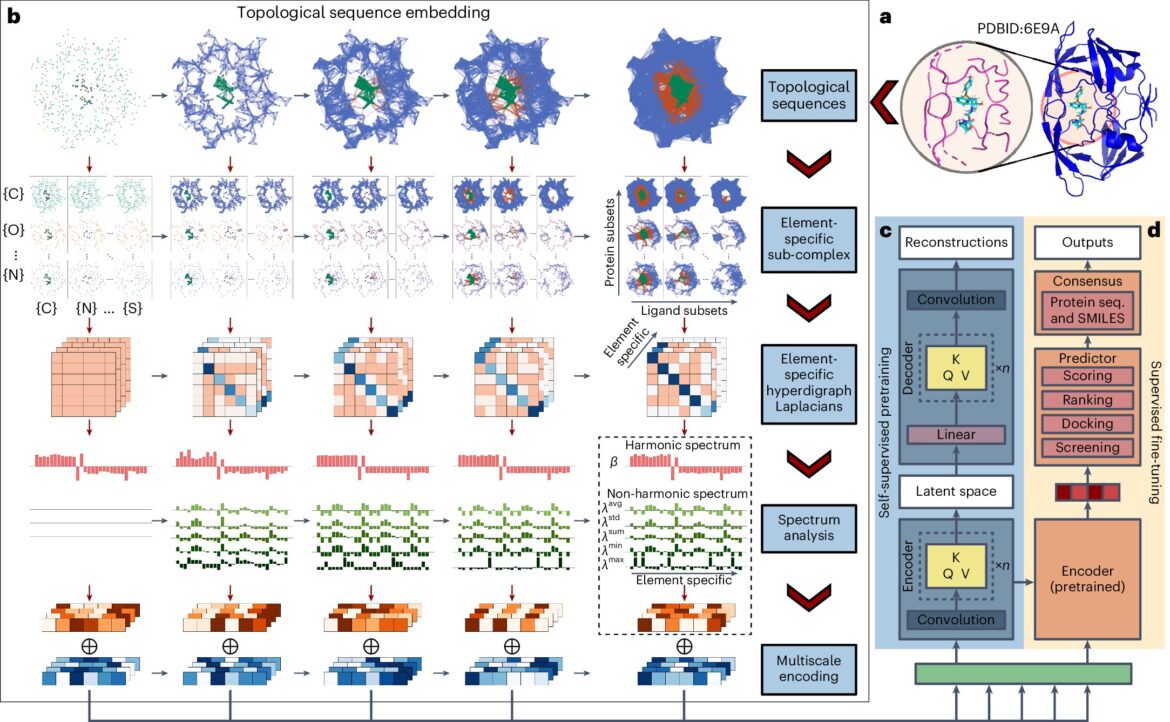
Illustration schématique du modèle TopoFormer global. Crédit: Intelligence des machines naturelles (2024). DOI : 10.1038/s42256-024-00855-1
Un nouveau programme basé sur l’IA permettra aux chercheurs d’intensifier leurs efforts de découverte de médicaments.
Le programme, appelé TopoFormer, a été développé par une équipe interdisciplinaire dirigée par Guowei Wei, professeur à la Michigan State University Research Foundation au département de mathématiques. TopoFormer traduit les informations tridimensionnelles sur les molécules en données que les modèles d'interaction médicamenteuse typiques basés sur l'IA peuvent utiliser, élargissant ainsi les capacités de ces modèles à prédire l'efficacité d'un médicament.
“Avec l'IA, vous pourriez rendre la découverte de médicaments plus rapide, plus efficace et moins chère”, a déclaré Wei, qui occupe également des postes au Département de biochimie et de biologie moléculaire et au Département de génie électrique et informatique.
Wei et son équipe ont publié un article sur leur travail dans la revue Intelligence des machines naturelles.
Instructions pour la structure
Aux États-Unis, le développement d'un seul médicament prend environ une décennie et coûte environ 2 milliards de dollars, a déclaré Wei. Tester le médicament avec des essais prend environ la moitié de ce temps, a-t-il ajouté, mais l'autre moitié est consacrée à la découverte d'un nouveau candidat thérapeutique à tester.
TopoFormer a le potentiel de réduire le temps de développement. Ce faisant, cela peut réduire les coûts de développement, ce qui pourrait faire baisser le prix du médicament pour les consommateurs en aval. Cela pourrait être particulièrement utile pour les maladies rares, car le nombre limité de patients oblige les sociétés pharmaceutiques à facturer davantage pour récupérer leurs coûts.
Bien que les chercheurs utilisent actuellement des modèles informatiques pour faciliter la découverte de médicaments, il existe des limites découlant de la myriade de variables du problème.
“Dans notre corps, nous avons plus de 20 000 protéines”, a déclaré Wei. “Lorsqu'une maladie apparaît, une ou plusieurs d'entre elles sont ciblées.”
La première étape consiste donc à déterminer quelle ou quelles protéines une maladie affecte. Ces protéines deviennent également des cibles pour les chercheurs qui souhaitent trouver des molécules capables de prévenir, minimiser ou contrecarrer les effets de la maladie.
“Quand j'ai une cible, j'essaie de trouver un grand nombre de médicaments potentiels pour cette cible particulière”, a déclaré Wei.
Une fois que les scientifiques savent quelles protéines cibler avec un médicament, ils peuvent saisir les séquences moléculaires de la protéine et des médicaments potentiels dans des modèles informatiques conventionnels. Les modèles prédisent la manière dont les médicaments et la cible interagiront, guidant ainsi les décisions sur les médicaments à développer et à tester dans les essais cliniques.
Bien que ces modèles puissent prédire certaines interactions en se basant uniquement sur la composition chimique du médicament et de la protéine, ils négligent également les interactions vitales qui proviennent de la forme moléculaire et de la structure tridimensionnelle ou 3D.
L'ibuprofène, découvert par les chimistes dans les années 1960, en est un exemple. Il existe deux molécules d’ibuprofène différentes qui partagent exactement la même séquence chimique mais ont des structures 3D légèrement différentes. Un seul arrangement est conçu de manière à pouvoir se lier aux protéines liées à la douleur et effacer un mal de tête.
“Les modèles d'apprentissage profond actuels ne peuvent pas tenir compte de la forme des médicaments ou des protéines pour prédire comment ils fonctionneront ensemble”, a déclaré Wei.
C'est là qu'intervient TopoFormer. Il s'agit d'un modèle de transformateur, le même type d'intelligence artificielle utilisé par le chatbot d'Open AI, ChatGPT (le GPT signifie « transformateur pré-entraîné génératif »).
Cela signifie que TopoFormer est formé pour lire les informations sous une forme et les transformer sous une autre forme. Dans ce cas, il prend des informations tridimensionnelles sur la façon dont les protéines et les médicaments interagissent en fonction de leurs formes et les recrée sous forme d’informations unidimensionnelles que les modèles actuels peuvent comprendre.
En fait, « Topo » signifie « Laplacien topologique », qui fait référence aux outils mathématiques inventés par Wei et son équipe pour convertir des structures 3D en séquences 1D.
Le nouveau modèle est formé sur des dizaines de milliers d'interactions protéine-médicament, où chaque interaction entre deux molécules est enregistrée sous forme de morceau de code, ou de « mot ». Les mots sont enchaînés pour créer une description du complexe médicament-protéine, créant ainsi un enregistrement de sa forme.
“De cette manière, vous avez de très nombreux mots liés ensemble comme une phrase”, a déclaré Wei.
Ces phrases peuvent ensuite être lues par d’autres modèles qui prédisent de nouvelles interactions médicamenteuses et leur donnent plus de contexte. Si un nouveau médicament est un livre, TopoFormer peut prendre une idée d’histoire approximative et la transformer en une intrigue à part entière, prête à être écrite.
Plus d'information:
Dong Chen et al, Transformateur structure-séquence activé par la topologie multi-échelle pour les prédictions d'interaction protéine-ligand, Intelligence des machines naturelles (2024). DOI : 10.1038/s42256-024-00855-1
Fourni par l'Université d'État du Michigan
Citation: Transformer la découverte de médicaments avec l'IA : un nouveau programme transforme les informations 3D en données que les modèles typiques peuvent utiliser (21 juin 2024) récupéré le 22 juin 2024 sur
Ce document est soumis au droit d'auteur. En dehors de toute utilisation équitable à des fins d'étude ou de recherche privée, aucune partie ne peut être reproduite sans autorisation écrite. Le contenu est fourni seulement pour information.