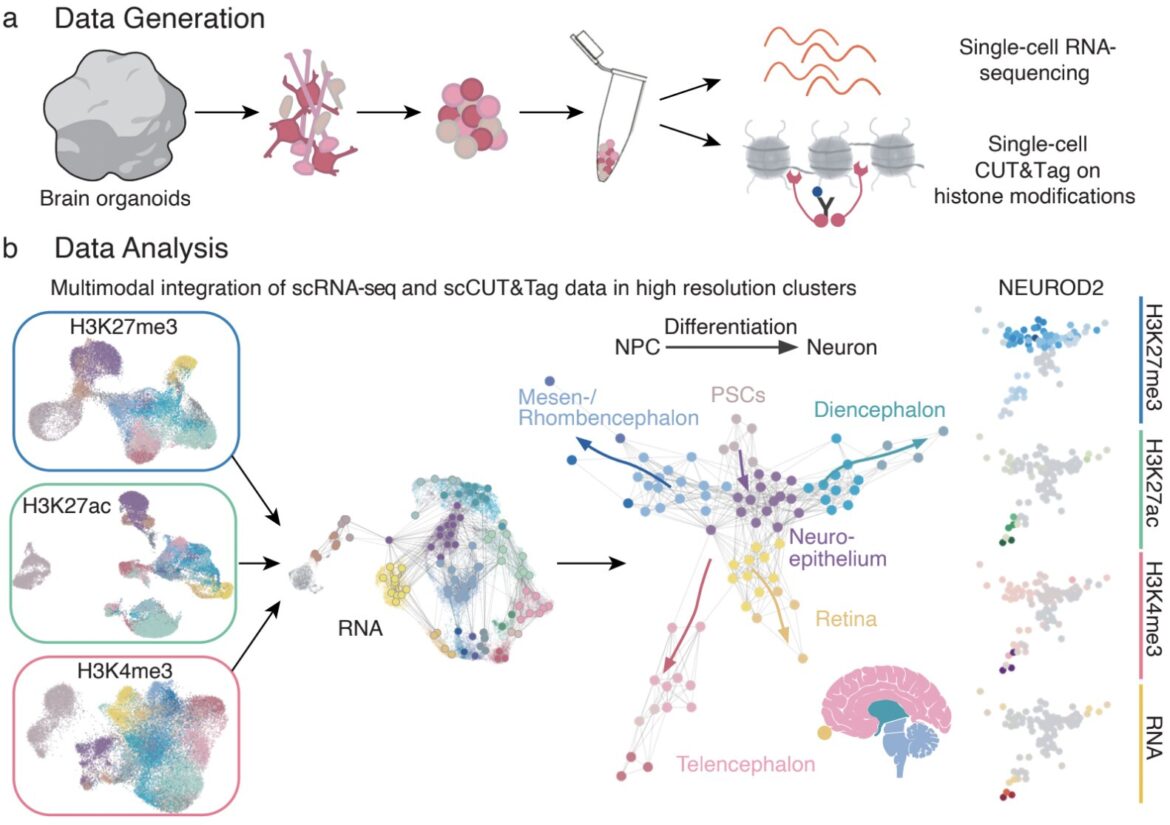
Analyse de l’atlas cellulaire développemental. a, Workflow pour obtenir l’expression de l’ARN monocellulaire (scRNA) et les données CUT&Tag pour trois modifications d’histones (H3K27me3, H3K4me3 et H3K27ac). b, En utilisant la correspondance bipartite MCMF et Cellrank2, nous avons dérivé des métacellules multiomiques et construit une disposition dirigée par la force qui montre les trajectoires développementales du neuroépithélium vers les principales régions cérébrales et les états de différenciation cellulaire. Expression de l’ARN et enrichissement des modifications d’histones tout au long du développement au niveau de NEUROD2. PSC, cellule souche pluripotente ; NPC, cellule précurseur neurale. Crédit : Zenk et al
Les cellules individuelles du corps humain se développent progressivement au fil du temps, pour finalement se spécialiser dans des fonctions spécifiques. Ce processus, appelé différenciation ou spécialisation cellulaire, est essentiel à la formation de populations cellulaires distinctes qui remplissent des fonctions différentes.
Des études antérieures suggèrent que le destin des cellules est également modulé par des mécanismes épigénétiques (c’est-à-dire des interactions entre gènes et facteurs environnementaux). Ces mécanismes épigénétiques se sont toutefois révélés jusqu’à présent difficiles à identifier.
Des chercheurs de l’ETH Zurich et de l’Institut Roche de biologie humaine ont récemment mené une étude visant à explorer les processus épigénétiques influençant les trajectoires de développement de cellules individuelles, en utilisant des organoïdes du cerveau et de la rétine humains dérivés de cellules souches pluripotentes.
Leur article, publié dans Neurosciences de la naturedécrit une carte de l’épigénome à l’échelle d’une cellule unique qui pourrait aider à l’étude de la détermination du destin des cellules humaines.
« Notre récent article dans Neurosciences de la nature « a été inspiré par le besoin de mieux comprendre les mécanismes épigénétiques qui régulent les décisions relatives au destin cellulaire au cours du développement du cerveau et de la rétine humaine », a déclaré Fides Zenk, co-auteur de l’article, à Medical Xpress.
« L’objectif principal était de créer une carte épigénomique unicellulaire complète capable de capturer les transitions des cellules souches pluripotentes aux cellules neurales différenciées. »
Zenk et ses collègues ont entrepris de créer une carte qui pourrait servir de « modèle » pour explorer comment la modification activatrice et répressive des histones orchestre la différenciation des cellules au cours de leur développement.
Organoïde cérébral de trois mois coloré au DAPI pour visualiser les noyaux. Crédit : Zenk et al.
Pour étudier les mécanismes épigénétiques à la base de la diversification des cellules humaines, les chercheurs ont mené des expériences sur des organoïdes du cerveau et de la rétine humains, des versions miniaturisées en trois dimensions (3D) d’organes humains créés en laboratoire, en utilisant des cellules souches pluripotentes.
« Nous avons réalisé un profilage unicellulaire de trois modifications clés des histones : H3K27ac (activatrice), H3K27me3 (répressive) et H3K4me3 (activatrice) », explique Zenk. « En utilisant des techniques telles que CUT&Tag (Cleavage Under Targets and Tagmentation) et le séquençage d’ARN unicellulaire (scRNA-seq), nous avons cartographié ces modifications à différents stades de développement, de la pluripotence aux états neuronaux différenciés. »
Grâce à des techniques épigénétiques, les chercheurs ont pu suivre l’évolution de ces trois modifications clés des histones au cours des différentes étapes de développement des cellules. Les observations recueillies au cours de leurs expériences leur ont permis d’identifier des « commutateurs » épigénétiques dynamiques qui régulent le destin des cellules individuelles.
« Nous avons identifié des commutateurs épigénétiques dynamiques qui régulent les décisions relatives au destin cellulaire et avons créé un atlas épigénomique unicellulaire du développement du cerveau et de la rétine humaine », a déclaré Zenk. « Nous avons découvert que la commutation des modifications répressives et activatrices des histones se produit avant les décisions relatives au destin cellulaire.
« De plus, nous avons démontré que l’élimination de H3K27me3 au stade du neuroectoderme perturbe la restriction du destin, conduisant à des identités cellulaires aberrantes. »
Les résultats de cette étude récente contribuent à la compréhension du développement des cellules humaines et de ses mécanismes épigénétiques sous-jacents. À l’avenir, les résultats de l’équipe et la carte qu’elle a créée pourraient contribuer à faire la lumière sur les bases épigénétiques de divers troubles du développement neurologique, tout en éclairant potentiellement la modélisation des maladies et le développement de nouvelles techniques de médecine régénératrice.
« Nos futurs projets de recherche incluent l’exploration plus approfondie des mécanismes par lesquels les modifications des histones interagissent pour réguler les décisions relatives au destin cellulaire et l’extension de notre cartographie épigénomique unicellulaire à d’autres modèles d’organoïdes et systèmes de développement », a ajouté Zenk.
« Nous souhaitons également étudier comment des modifications épigénétiques spécifiques contribuent aux états pathologiques et explorer des interventions thérapeutiques qui pourraient moduler ces marques épigénétiques pour restaurer la fonction cellulaire normale. »
Plus d’information:
Fides Zenk et al, Reconstruction épigénomique unicellulaire des trajectoires de développement à partir de la pluripotence dans les systèmes organoïdes neuronaux humains, Neurosciences de la nature (2024). DOI: 10.1038/s41593-024-01652-0
© 2024 Réseau Science X
Citation:Une étude identifie des « commutateurs » épigénétiques qui régulent les trajectoires de développement des cellules individuelles (2024, 15 juillet) récupéré le 15 juillet 2024 à partir de
Ce document est soumis au droit d’auteur. En dehors de toute utilisation équitable à des fins d’étude ou de recherche privée, aucune partie ne peut être reproduite sans autorisation écrite. Le contenu est fourni à titre d’information uniquement.