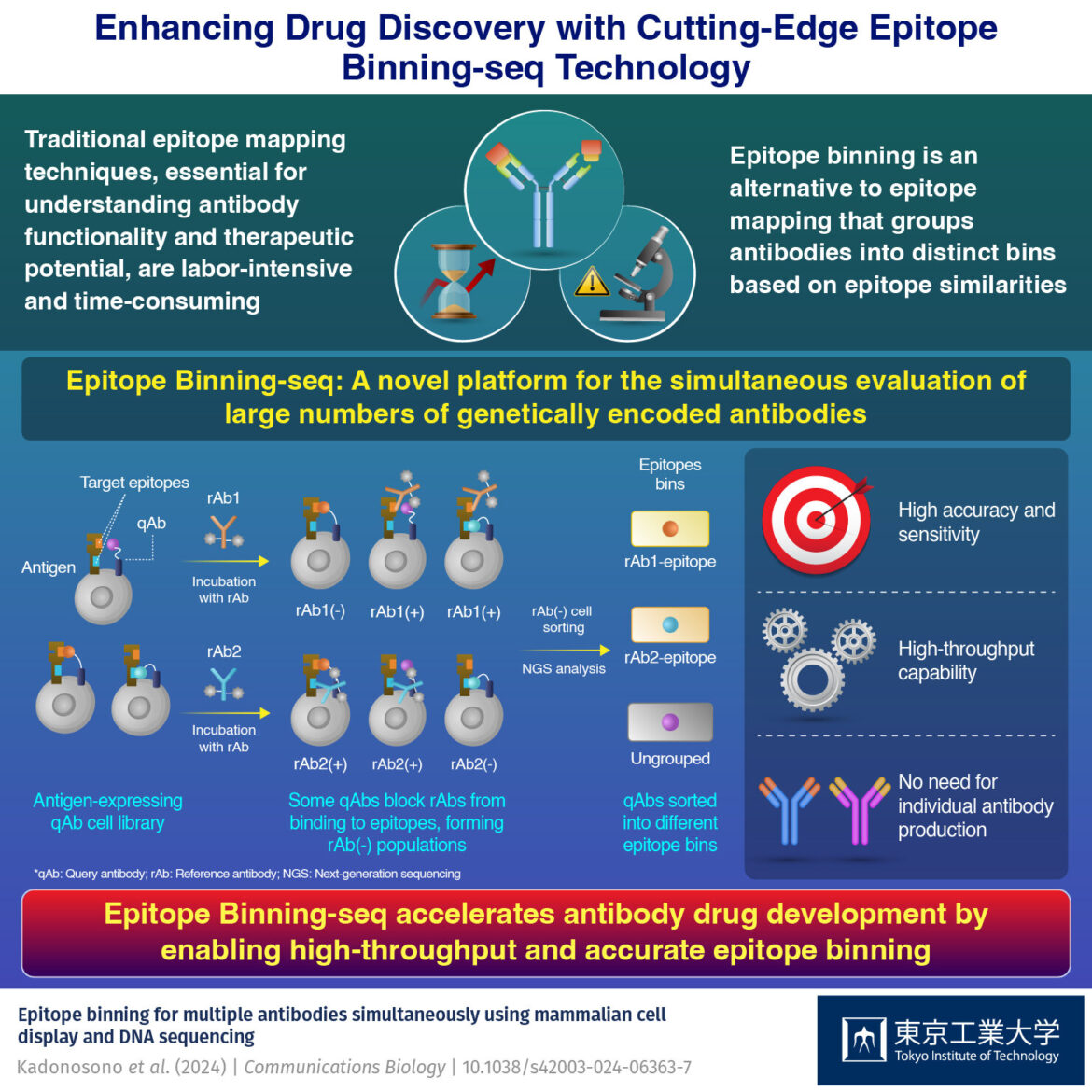
La nouvelle technologie Epitope Binning-seq rationalise la cartographie des épitopes d'anticorps et permet l'évaluation simultanée d'un grand nombre d'anticorps génétiquement codés, en les regroupant dans des groupes d'épitopes distincts avec une précision et une sensibilité élevées. Crédit : Tokyo Tech
Les anticorps monoclonaux sont des agents thérapeutiques essentiels en raison de leur spécificité élevée et de leur affinité de liaison pour les épitopes (les sites précis où un anticorps s'attache à un antigène, déclenchant une réponse immunitaire). Puisque l’efficacité thérapeutique des anticorps est étroitement liée à leurs épitopes cibles, la caractérisation des épitopes est essentielle pour comprendre la fonctionnalité des anticorps.
Cependant, les méthodes traditionnelles utilisées pour étudier les épitopes sont lentes et demandent beaucoup de travail. Il existe donc un besoin urgent d’approches plus efficaces permettant de cartographier les épitopes avec précision et rapidité, accélérant ainsi le développement de thérapies de nouvelle génération.
Pour combler cette lacune, des chercheurs de l’Institut de technologie de Tokyo se sont lancés dans des recherches révolutionnaires, aboutissant au développement d’une nouvelle plateforme appelée Epitope Binning-seq. Leurs conclusions ont été publiées le 28 mai 2024 dans Biologie des communications. Epitope Binning-seq évalue la similarité des épitopes à l'aide d'anticorps de requête (qAb) génétiquement codés sur des cellules exprimant l'antigène et du séquençage de nouvelle génération (NGS), permettant l'évaluation simultanée de plusieurs qAb sans purification individuelle.
Dans cette étude, des anticorps de requête (qAbs) affichés à la surface des cellules exprimant HER2 ont été utilisés. En introduisant des anticorps de référence (rAb) marqués par fluorescence et en utilisant une analyse par cytométrie en flux, les chercheurs ont pu distinguer les cellules où les qAb masquaient les épitopes et les cellules où ils ne le faisaient pas.
Certains qAb ont efficacement bloqué la liaison du rAb à l'antigène, ce qui a donné lieu à une population de cellules sans liaison au rAb, appelée population rAb-négative. À l’inverse, d’autres qAb ont permis au rAb de se lier à l’antigène, créant ainsi une population de cellules avec une liaison au rAb, connue sous le nom de population rAb-positive.
Donnant de plus amples informations sur leur étude, le professeur agrégé Tetsuya Kadonosono, l'auteur correspondant de l'étude, déclare : « Le comportement de liaison différentiel a servi de base pour évaluer la similarité des épitopes entre différents anticorps. Nous avons effectué une analyse NGS sur les populations triées négatives pour les anticorps rAb. identifiez et regroupez les qAb similaires dans des bacs d'épitopes. Cette approche globale a permis une analyse efficace d'un grand nombre d'anticorps et une classification basée sur leur spécificité d'épitope.
Les résultats étaient plutôt prometteurs. Epitope Binning-seq a classé avec précision les anticorps dans des groupes d'épitopes distincts, fournissant ainsi des informations précieuses sur leurs modèles de liaison. Dans des expériences utilisant des anticorps modèles pertuzumab et trastuzumab, la méthode a identifié et enrichi avec précision des qAb spécifiques, détectant même des clones à des abondances initiales très faibles.
La plateforme a classé 14 qAb dans des groupes d’épitopes corrects, avec une grande précision. Ces résultats mettent en valeur le potentiel d'Epitope Binning-seq à rationaliser le développement précoce de médicaments à base d'anticorps en évaluant simultanément des millions de qAb, accélérant ainsi l'identification de candidats thérapeutiques prometteurs.
Les avantages de cette plateforme sont importants. Epitope Binning-seq rationalise la comparaison des épitopes, permettant une identification rapide des anticorps présentant des modèles de liaison similaires, ce qui pourrait faire progresser les thérapies ciblées et efficaces basées sur les anticorps. L'évaluation à grande échelle de la méthode peut transformer la caractérisation des anticorps, permettant d'évaluer des millions de qAb simultanément.
« Le développement d'Epitope Binning-seq représente une avancée majeure dans la découverte de médicaments à base d'anticorps. En fournissant une méthode rapide, complète et rentable pour évaluer la similarité des épitopes d'anticorps, cette nouvelle plateforme surmonte les limites des techniques traditionnelles d'analyse d'épitopes », conclut Kadonosono. .
Les résultats prometteurs de cette étude mettent en valeur le potentiel de cette méthode pour transformer le développement précoce de médicaments à base d’anticorps et la maturation de l’affinité des anticorps, ouvrant ainsi la voie à des stratégies thérapeutiques plus efficaces à l’avenir.
Plus d'information:
Ning Lin et al, Regroupement d'épitopes pour plusieurs anticorps simultanément à l'aide de l'affichage de cellules de mammifères et du séquençage de l'ADN, Biologie des communications (2024). DOI : 10.1038/s42003-024-06363-7
Fourni par l'Institut de technologie de Tokyo
Citation: Une nouvelle plateforme rationalise la comparaison des épitopes dans la recherche sur les anticorps (10 juin 2024) récupéré le 10 juin 2024 sur
Ce document est soumis au droit d'auteur. En dehors de toute utilisation équitable à des fins d'étude ou de recherche privée, aucune partie ne peut être reproduite sans autorisation écrite. Le contenu est fourni seulement pour information.