Crédit: Journal américain de génétique humaine (2024). DOI : 10.1016/j.ajhg.2024.06.011
Des chercheurs de l’hôpital pour enfants de Philadelphie (CHOP) et de la Perelman School of Medicine de l’université de Pennsylvanie (Penn Medicine) ont développé des données destinées à améliorer le diagnostic du syndrome d’Alagille, une maladie hépatique héréditaire complexe. Ces résultats contribueront à réduire la classification des variantes de signification incertaine (VOUS) pour les variantes faux-sens, des changements génétiques altérant les protéines, qui ont des implications pour d’autres maladies. Les résultats ont été publiés aujourd’hui par le Journal américain de génétique humaine.
Le syndrome d’Alagille, également connu sous le nom de syndrome d’Alagille-Watson, de dysplasie artério-hépatique et de chéloïdite biliaire syndromique, est une maladie hépatique héréditaire qui affecte également le cœur, les yeux, les os, les reins, le système vasculaire et d’autres organes, avec une gravité variable. Le syndrome d’Alagille est principalement causé par des variantes du gène Jagged1 (JAG1), qui contribue à la production d’une protéine qui contrôle la croissance et le développement des cellules en différents types de tissus et d’organes lorsqu’elles fonctionnent correctement.
De nombreuses modifications génétiques entraînant un raccourcissement des protéines peuvent être facilement identifiées comme étant à l’origine de la maladie et conduire à un diagnostic correct du syndrome d’Alagille, permettant ainsi aux enfants de recevoir un traitement approprié. Cependant, jusqu’à 85 % des variantes faux-sens JAG1 signalées (mutations dans lesquelles un acide aminé est remplacé par un autre qui n’est généralement pas à cette position dans le gène) ont des classifications incertaines ou contradictoires, ce qui signifie que les patients atteints du syndrome d’Alagille pourraient passer inaperçus.
« Nous voulions nous concentrer sur les exons où ces variantes faux-sens étaient les plus susceptibles de se produire et analyser les variantes qui pourraient être comparées à celles dont nous savons qu’elles sont responsables de la maladie », a déclaré la première auteure de l’étude, Melissa Gilbert, Ph.D., professeure adjointe de recherche en pathologie et médecine de laboratoire à Penn Medicine et CHOP. « En utilisant diverses méthodes, nous avons développé un test à haut débit et avons pu réduire l’incertitude lorsqu’il s’agit de classer ces variantes faux-sens et d’améliorer notre capacité à fournir un diagnostic plus précis du syndrome d’Alagille. »
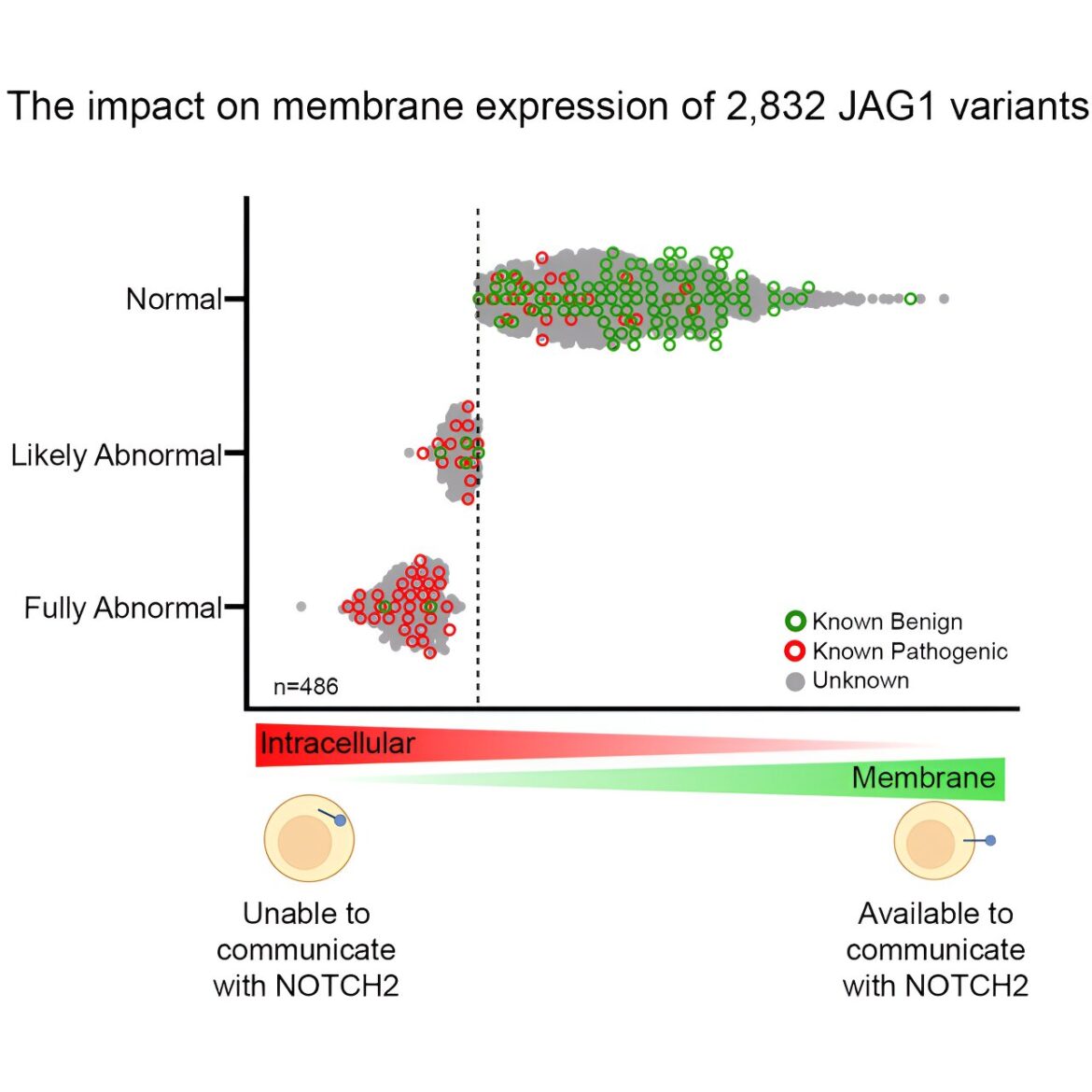
Chaque gène contient des exons, qui sont les régions codantes qui fournissent des instructions pour la fabrication de protéines spécifiques. Dans cette étude, les chercheurs ont généré une bibliothèque de 2 832 variantes de nucléotides JAG1 dans les exons 1 à 7, une région du gène présentant un nombre élevé de variantes faux-sens signalées.
En utilisant un test à haut débit pour mesurer l’expression de la membrane JAG1 et en calibrant à l’aide d’un ensemble distinct de variantes de contrôle, les chercheurs ont caractérisé 486 variantes comme fonctionnellement anormales, dont 439 étaient des variantes faux-sens.
Ces informations ont ensuite été comparées à celles de 144 variants incertains signalés chez des patients soumis à des tests cliniques ou de recherche. Parmi ces variants, 27 présentaient une expression membranaire fonctionnellement anormale de JAG1 et, à la suite de ces résultats, 26 ont été reclassés comme probablement pathogènes ou responsables du syndrome d’Alagille.
« La méthode que nous avons utilisée pour re-caractériser ces variants a des implications pour d’autres facteurs génétiques de maladies rares et complexes », a déclaré Nancy B. Spinner, Ph.D., auteure principale de l’étude et chef de la division de diagnostic génomique au CHOP. « Dans un avenir proche, nous espérons que cet ensemble de données pourra fournir aux conseillers en génétique et aux cliniciens une capacité améliorée de classification de ces variants lorsqu’ils sont détectés pour la première fois chez des patients atteints du syndrome d’Alagille et réduire l’incertitude et l’anxiété auxquelles sont confrontées les familles de patients confrontés à cette maladie difficile. »
Les objectifs futurs des chercheurs incluent l’extension de ces travaux pour résoudre toutes les variantes du JAG1, l’extension à d’autres gènes de maladies et le développement de protocoles pour intégrer ces données dans l’interprétation des variantes dans le laboratoire de diagnostic clinique du CHOP. La résolution du VOUS sur tous les gènes médicalement significatifs est une priorité déclarée du National Human Genome Research Institute.
Plus d’information:
Melissa A. Gilbert et al., La caractérisation fonctionnelle de 2 832 variantes de JAG1 appuie la reclassification du syndrome d’Alagille et améliore les indications pour l’interprétation des variantes cliniques, Journal américain de génétique humaine (2024). DOI : 10.1016/j.ajhg.2024.06.011
Fourni par l’hôpital pour enfants de Philadelphie
Citation: La reclassification des variantes à haut débit facilite le diagnostic du syndrome d’Alagille (2024, 22 juillet) récupéré le 22 juillet 2024 à partir de
Ce document est soumis au droit d’auteur. En dehors de toute utilisation équitable à des fins d’étude ou de recherche privée, aucune partie ne peut être reproduite sans autorisation écrite. Le contenu est fourni à titre d’information uniquement.