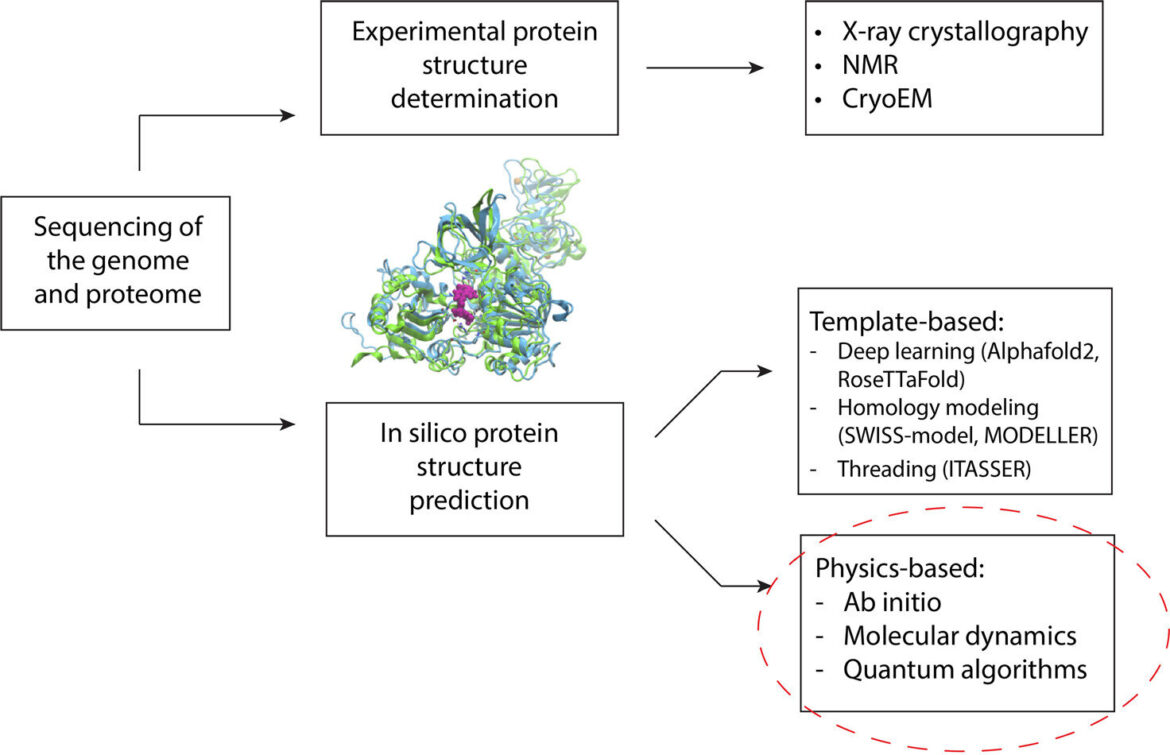
Présentation du pipeline PSP. Après le séquençage génomique, la séquence primaire d’acides aminés est déterminée. La méthode expérimentale commence alors par exprimer cette protéine en modifiant génétiquement un autre organisme avec cette nouvelle séquence. Cet organisme traduira ensuite ces protéines, et la nouvelle protéine d’intérêt pourra être isolée, purifiée, puis résolue par cristallographie aux rayons X, RMN ou CryoEM. Les méthodes in silico, en revanche, prennent simplement la séquence d'acides aminés primaires comme entrée et la structure est prédite soit par une méthode basée sur la physique (dans laquelle la biophysique sous-jacente est simulée d'une manière ou d'une autre) ou par une méthode basée sur un modèle (dans laquelle des algorithmes d'apprentissage automatique prédire les structures en fonction des modèles trouvés dans un ensemble d'apprentissage de modèles expérimentaux). La méthode que nous adoptons dans ce travail entre dans la catégorie des algorithmes basés sur la physique. À titre d’exemple illustré, un modèle in silico et une structure cristalline aux rayons X de l’hélicase SARS-CoV2 NSP13 (PDB : 7NN0) sont superposés, ainsi qu’un inhibiteur connu ancré (coloré en magenta). Crédit: Journal de théorie chimique et de calcul (2024). DOI : 10.1021/acs.jctc.4c00067
Des chercheurs de la Cleveland Clinic et d'IBM ont récemment publié leurs résultats dans la revue Journal de théorie chimique et de calcul cela pourrait jeter les bases de l’application des méthodes informatiques quantiques à la prédiction de la structure des protéines.
Pendant des décennies, les chercheurs ont exploité des approches informatiques pour prédire les structures des protéines. Une protéine se replie dans une structure qui détermine son fonctionnement et se lie à d’autres molécules du corps. Ces structures déterminent de nombreux aspects de la santé humaine et des maladies.
En prédisant avec précision la structure d’une protéine, les chercheurs peuvent mieux comprendre comment les maladies se propagent et ainsi développer des thérapies efficaces. Bryan Raubenolt, boursier postdoctoral de la Cleveland Clinic, Ph.D. et le chercheur IBM Hakan Doga, Ph.D. a dirigé une équipe pour découvrir comment l’informatique quantique peut améliorer les méthodes actuelles.
Ces dernières années, les techniques d’apprentissage automatique ont fait des progrès significatifs dans la prédiction de la structure des protéines. Ces méthodes s'appuient sur des données d'entraînement (une base de données de structures protéiques déterminées expérimentalement) pour faire des prédictions. Cela signifie qu’ils sont limités par le nombre de protéines qu’on leur a appris à reconnaître. Cela peut conduire à des niveaux de précision inférieurs lorsque les programmes/algorithmes rencontrent une protéine mutée ou très différente de celles sur lesquelles ils ont été formés, ce qui est courant dans les troubles génétiques.
La méthode alternative consiste à simuler la physique du repliement des protéines. Les simulations permettent aux chercheurs d'examiner les différentes formes possibles d'une protéine donnée et de trouver la plus stable. La forme la plus stable est essentielle à la conception de médicaments.
Le défi est que ces simulations sont quasiment impossibles sur un ordinateur classique, au-delà d’une certaine taille de protéine. D'une certaine manière, augmenter la taille de la protéine cible est comparable à l'augmentation des dimensions d'un Rubik's cube. Pour une petite protéine contenant 100 acides aminés, un ordinateur classique aurait besoin d'un temps égal à l'âge de l'univers pour rechercher de manière exhaustive tous les résultats possibles, explique le Dr Raubenolt.
Pour aider à surmonter ces limites, l’équipe de recherche a appliqué un mélange de méthodes informatiques quantiques et classiques. Ce cadre pourrait permettre aux algorithmes quantiques d'aborder les domaines qui représentent un défi pour l'informatique classique de pointe, notamment la taille des protéines, le désordre intrinsèque, les mutations et la physique impliquée dans le repliement des protéines. Le cadre a été validé en prédisant avec précision le repliement d'un petit fragment d'une protéine du virus Zika sur un ordinateur quantique, par rapport aux méthodes classiques de pointe.
Les premiers résultats du cadre hybride quantique-classique ont surpassé à la fois une méthode basée sur la physique classique et AlphaFold2. Bien que ce dernier soit conçu pour fonctionner au mieux avec des protéines plus grosses, il démontre néanmoins la capacité de ce cadre à créer des modèles précis sans s'appuyer directement sur des données d'entraînement substantielles.
Les chercheurs ont utilisé un algorithme quantique pour modéliser d'abord la conformation d'énergie la plus basse pour le squelette du fragment, ce qui constitue généralement l'étape de calcul la plus exigeante en termes de calcul. Des approches classiques ont ensuite été utilisées pour convertir les résultats obtenus à partir de l’ordinateur quantique, reconstruire la protéine avec ses chaînes latérales et effectuer le raffinement final de la structure avec les champs de force de la mécanique moléculaire classique.
Le projet montre l'une des façons dont les problèmes peuvent être déconstruits en parties, avec des méthodes informatiques quantiques s'attaquant à certaines parties et à l'informatique classique d'autres, pour une précision accrue.
“L'un des aspects les plus uniques de ce projet est le nombre de disciplines impliquées”, explique le Dr Raubenolt. « L'expertise de notre équipe s'étend de la biologie et de la chimie computationnelles, de la biologie structurale, de l'ingénierie logicielle et de l'automatisation, à la physique atomique et nucléaire expérimentale, aux mathématiques et, bien sûr, à l'informatique quantique et à la conception d'algorithmes. Il a fallu les connaissances de chacun de ces domaines pour créer un cadre informatique capable d'imiter l'un des processus les plus importants de la vie humaine.
La combinaison de méthodes informatiques classiques et quantiques par l'équipe constitue une étape essentielle pour faire progresser notre compréhension des structures protéiques et de leur impact sur notre capacité à traiter et à prévenir les maladies. L’équipe prévoit de continuer à développer et à optimiser des algorithmes quantiques capables de prédire la structure de protéines plus grandes et plus sophistiquées.
“Ce travail constitue une étape importante dans l'exploration des domaines dans lesquels les capacités de calcul quantique pourraient démontrer leurs atouts en matière de prédiction de la structure des protéines”, déclare le Dr Doga. “Notre objectif est de concevoir des algorithmes quantiques capables de prédire les structures des protéines de la manière la plus réaliste possible.”
Plus d'information:
Hakan Doga et al, Une perspective sur la prédiction de la structure des protéines à l'aide d'ordinateurs quantiques, Journal de théorie chimique et de calcul (2024). DOI : 10.1021/acs.jctc.4c00067
Fourni par la clinique de Cleveland
Citation: Des chercheurs appliquent des méthodes d'informatique quantique à la prédiction de la structure des protéines (29 mai 2024) récupéré le 29 mai 2024 sur
Ce document est soumis au droit d'auteur. En dehors de toute utilisation équitable à des fins d'étude ou de recherche privée, aucune partie ne peut être reproduite sans autorisation écrite. Le contenu est fourni seulement pour information.