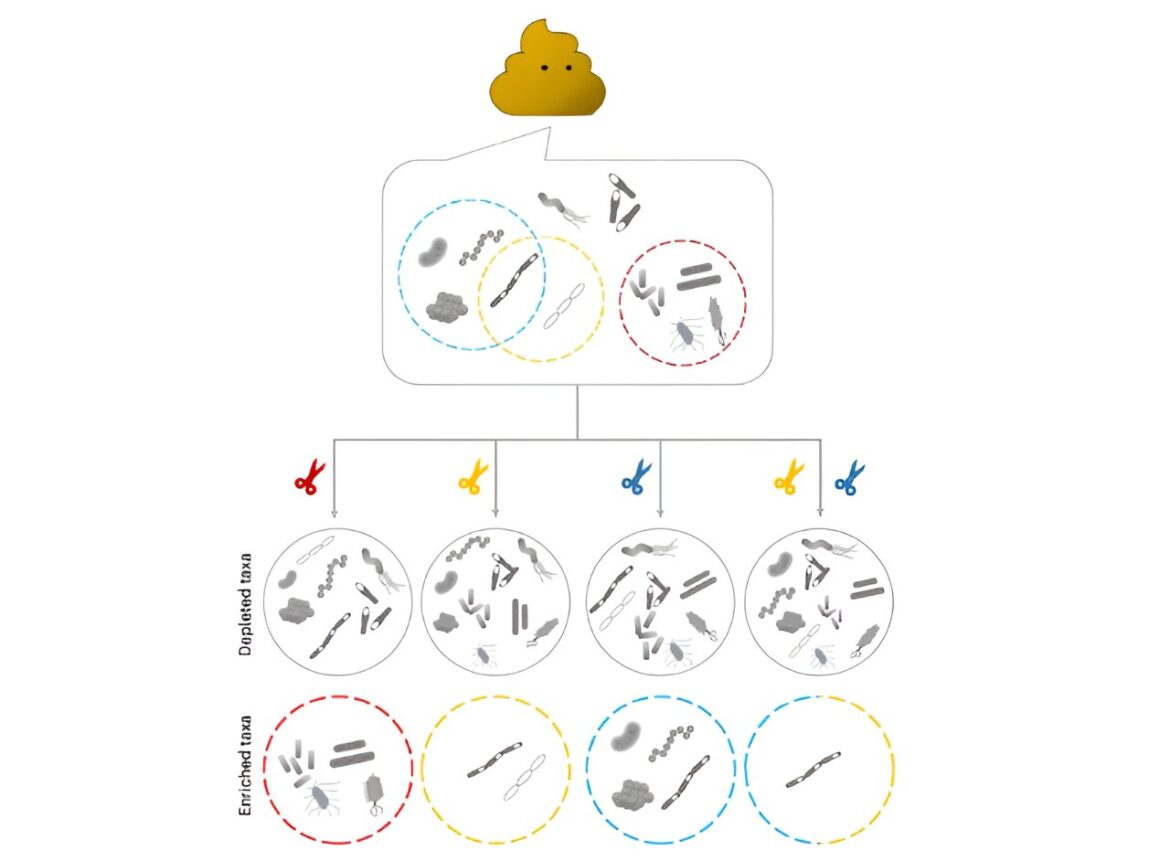
mEnrich-seq peut être utilisé pour examiner diverses bactéries d’intérêt à partir du même échantillon de microbiome. Crédit : École de médecine Icahn du Mont Sinaï
Dans une étude historique publiée dans la revue Méthodes naturelles, des chercheurs de l'École de médecine Icahn du Mont Sinaï ont dévoilé mEnrich-seq, une méthode innovante conçue pour améliorer considérablement la spécificité et l'efficacité de la recherche sur les microbiomes, les communautés complexes de micro-organismes qui habitent le corps humain. L'article s'intitule « mEnrich-seq : séquençage d'enrichissement guidé par méthylation de taxons bactériens d'intérêt à partir du microbiome ».
Libérer le monde microbien avec mEnrich-seq
Les microbiomes jouent un rôle crucial dans la santé humaine. Un déséquilibre ou une diminution de la variété des microbes dans notre corps peut entraîner un risque accru de plusieurs maladies. Cependant, dans de nombreuses applications du microbiome, l’accent est mis sur l’étude de types spécifiques de bactéries dans un échantillon, plutôt que sur chaque type présent. Par exemple, lorsqu’ils étudient les maladies infectieuses, les chercheurs peuvent s’intéresser uniquement à quelques bactéries intestinales nocives, mais celles-ci sont mélangées à de nombreuses autres bactéries.
“Imaginez que vous êtes un scientifique qui doit étudier un type particulier de bactérie dans un environnement complexe. C'est comme essayer de trouver une aiguille dans une grosse botte de foin”, a déclaré Gang Fang, Ph.D., professeur de génétique et de sciences génomiques et l'auteur principal de l'étude. « mEnrich-seq donne essentiellement aux chercheurs une « pince à épiler intelligente » pour récupérer l'aiguille qui les intéresse.
Une fois extraits par la « pince intelligente », les chercheurs peuvent assembler le(s) génome(s) des bactéries ciblées, facilitant ainsi l'étude de diverses questions biomédicales les concernant.
Cette nouvelle stratégie comble une lacune technologique critique, car auparavant les chercheurs devaient isoler des souches bactériennes spécifiques d'un échantillon donné en utilisant des milieux de culture qui cultivent sélectivement la bactérie spécifique, un processus long qui fonctionne pour certaines bactéries, mais pas pour d'autres. mEnrich-seq, en revanche, peut récupérer directement le(s) génome(s) des bactéries d’intérêt à partir de l’échantillon de microbiome sans culture.
mEnrich-seq distingue efficacement les bactéries d'intérêt du vaste contexte en exploitant les « codes secrets » écrits sur l'ADN bactérien que les bactéries utilisent naturellement pour se différencier les unes des autres dans le cadre de leur système immunitaire natif.
Transformer la recherche et les soins de santé
L’avènement de mEnrich-seq ouvre de nouveaux horizons dans divers domaines :
- Rentabilité : il offre une approche plus économique de la recherche sur le microbiome, particulièrement bénéfique dans les études à grande échelle où les ressources peuvent être limitées.
- Large applicabilité : la méthode peut se concentrer sur un large éventail de bactéries, ce qui en fait un outil polyvalent pour la recherche et les applications cliniques.
- Percées médicales : en permettant une recherche plus ciblée, mEnrich-seq pourrait accélérer le développement de nouveaux outils de diagnostic et de nouveaux traitements.
“L'un des aspects les plus intéressants de mEnrich-seq est sa capacité à découvrir des détails précédemment manqués, comme les gènes de résistance aux antibiotiques que les méthodes de séquençage traditionnelles ne pouvaient pas détecter en raison d'un manque de sensibilité”, a ajouté le Dr Fang. “Cela pourrait constituer une avancée significative dans la lutte contre le problème mondial de la résistance aux antibiotiques.”
En effet, comme l'a démontré l'une des trois applications de cette étude, les auteurs ont utilisé mEnrich-seq pour reconstruire directement les génomes pathogènes d'E. coli à partir d'échantillons d'urine de patients atteints d'infections des voies urinaires, ce qui a permis une analyse complète des gènes de résistance aux antibiotiques dans chaque génome. .
Dans une autre application, les auteurs ont utilisé mEnrich-seq pour construire sélectivement les génomes d'Akkermansia muciniphila, une bactérie dont il a été démontré qu'elle présente des avantages dans l'obésité et le diabète, entre autres maladies, ainsi qu'une réponse à l'immunothérapie anticancéreuse.
Cette bactérie est difficile à cultiver, c'est pourquoi mEnrich-seq peut être un outil utile pour reconstruire son génome de manière indépendante de la culture, sensible et rentable, ce qui peut faciliter des études d'association à plus grande échelle avec différentes maladies humaines.
L'avenir de mEnrich-seq
Pour l’avenir, l’équipe a des projets ambitieux pour mEnrich-seq. Ils visent à affiner la méthode pour améliorer encore son efficacité et élargir sa gamme d’applications. Des collaborations avec des cliniciens et des professionnels de la santé sont également en cours pour valider l'utilité de la méthode dans des contextes réels.
« Nous envisageons mEnrich-seq comme un outil sensible et polyvalent dans l'avenir des études sur le microbiome et des applications cliniques », a déclaré le Dr Fang.
Plus d'information:
mEnrich-seq : séquençage d'enrichissement guidé par méthylation de taxons bactériens d'intérêt à partir du microbiome, Méthodes naturelles (2024). DOI : 10.1038/s41592-023-02125-1 www.nature.com/articles/s41592-023-02125-1
Fourni par l'Hôpital Mont Sinaï
Citation: La « pince à épiler » génomique inaugure une nouvelle ère de précision dans la recherche sur le microbiome (4 janvier 2024) récupéré le 4 janvier 2024 sur
Ce document est soumis au droit d'auteur. En dehors de toute utilisation équitable à des fins d'étude ou de recherche privée, aucune partie ne peut être reproduite sans autorisation écrite. Le contenu est fourni seulement pour information.