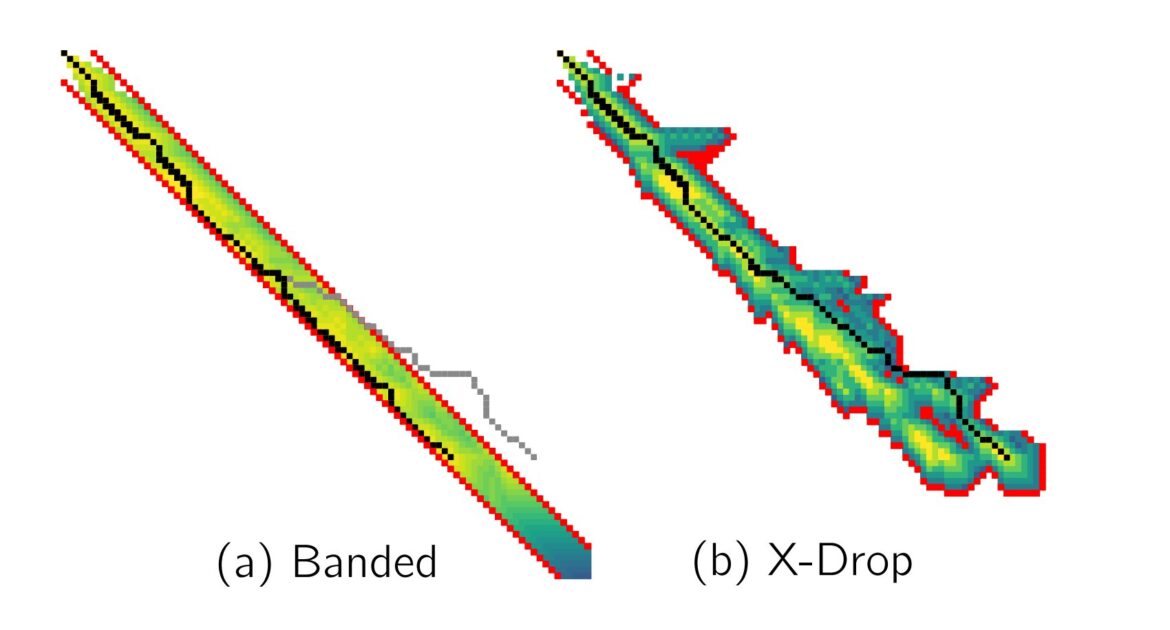
À gauche, l’alignement est obligé de rester dans la zone baguée quel que soit le score, manquant ainsi l’alignement optimal (gris). À droite, lorsque le score (jaune-bleu) 𝑋 descend en dessous du meilleur score actuel, la recherche prend fin (limite rouge) et l’alignement optimal (noir) est renvoyé. Crédit: arXiv (2023). DOI : 10.48550/arxiv.2304.08662
Un accélérateur matériel initialement développé pour les opérations d’intelligence artificielle accélère avec succès l’alignement des molécules de protéines et d’ADN, rendant le processus jusqu’à 10 fois plus rapide que les méthodes de pointe.
Cette approche peut rendre plus efficace l’alignement des séquences protéiques et de l’ADN pour l’assemblage du génome, ce qui constitue un problème fondamental en biologie computationnelle.
Giulia Guidi, professeur adjoint d’informatique au Cornell Ann S. Bowers College of Computing and Information Science, a mené une étude pour tester les performances de l’accélérateur, appelé unité de traitement de l’intelligence (IPU), en utilisant les données existantes sur les séquences d’ADN et de protéines. L’IPU accélère le processus d’alignement en fournissant plus de mémoire pour accélérer le mouvement des données, un problème courant.
“L’alignement des séquences est une partie extrêmement importante et gourmande en calcul de pratiquement toute charge de travail en biologie computationnelle”, a déclaré Guidi. “C’est extrêmement courant et c’est généralement l’un des goulots d’étranglement du calcul.”
L’étude, « Space Efficient Sequence Alignment for SRAM-Based Computing : X-Drop on the Graphcore IPU », sera présentée par le co-premier auteur Luk Burchard, ancien chercheur invité à Cornell et doctorant au Simula Research Laboratory, au Conférence Supercomputing2023, 14 novembre. Max Xiaohang Zhao, également ancien chercheur invité à Cornell, maintenant à la Charité Universitätsmedizin, est également co-premier auteur.
Dans ses recherches, Guidi souhaite aider les scientifiques à résoudre des problèmes qu’ils n’ont pas encore tentés car ils nécessitent une grande puissance de calcul. Ces problèmes complexes nécessitent des calculs à grande échelle : des assemblages de processeurs, de mémoire, de réseaux et de stockage de données capables de gérer de grandes tâches informatiques.
L’alignement de séquences d’ADN ou de protéines fait partie de ces problèmes complexes. Lorsqu’ils séquencent un génome, les biologistes se retrouvent avec des milliers, voire des millions de courtes séquences d’ADN qu’il faut assembler comme un puzzle. Ils utilisent un algorithme pour identifier les paires de séquences qui se chevauchent, puis relient les paires.
Au cours de la dernière décennie, les scientifiques se sont tournés vers les unités de traitement graphique (GPU), initialement développées pour accélérer le rendu graphique dans les jeux vidéo, afin d’accélérer l’alignement des séquences en exécutant des calculs en parallèle. Avec le développement des IPU pour les applications d’IA, Guidi et ses collègues voulaient savoir s’ils pouvaient exploiter les nouveaux accélérateurs pour résoudre ce problème.
“Le besoin de calcul à grande échelle augmente dans de nombreux domaines scientifiques, car nous sommes bien meilleurs que jamais dans la génération de données”, a déclaré Guidi. “L’informatique parallèle est passée du statut de luxe à celui de quelque chose de non négociable.”
Les IPU ont attiré Guidi car elles disposent d’une bande passante importante sur l’appareil pour le transfert de données et peuvent gérer des charges de travail inégales et imprévisibles. X-Drop, un algorithme populaire pour aligner des séquences, a un modèle de calcul très irrégulier. Lorsque deux séquences correspondent, l’algorithme nécessite beaucoup de calculs pour déterminer le bon alignement, mais lorsqu’elles ne correspondent pas, l’algorithme s’arrête. Les GPU ont du mal à gérer ce type de calcul irrégulier, mais l’IPU a excellé.
Lorsque le groupe de Guidi a assemblé des séquences d’organismes modèles E. coli et C. elegans à l’aide de l’IPU, ils ont obtenu des performances 10 fois plus rapides par rapport à un GPU, qui passe trop de temps à transférer des données inutilement, et des performances 4,65 fois plus rapides. qu’une unité centrale de traitement (CPU) sur un superordinateur.
Actuellement, ce qui limite la taille des génomes que les scientifiques peuvent traiter, c’est le nombre de périphériques IPU et GPU disponibles, ainsi que la bande passante pour le transfert de données entre le processeur hôte et l’accélérateur matériel. Il y a beaucoup de mémoire sur l’IPU, mais le transfert des données depuis l’hôte provoque un goulot d’étranglement majeur.
L’équipe a contribué à résoudre ce problème en réduisant de 55 fois l’empreinte mémoire de l’algorithme X-Drop. Cela lui a permis de fonctionner sur l’IPU et de réduire la quantité de données transférées depuis le CPU. En conséquence, le système a pu effectuer des comparaisons plus importantes et effectuer davantage de comparaisons de séquences sur l’UIP, ce qui a contribué à équilibrer la charge de travail inégale.
“Vous pouvez exploiter la bande passante mémoire élevée de l’IPU, ce qui vous permet d’accélérer l’ensemble du traitement”, a déclaré Guidi.
Si les fournisseurs parviennent à mettre à niveau le processus de transfert de données entre le processeur et l’IPU, ainsi qu’à améliorer l’écosystème logiciel, Guidi espère pouvoir traiter des génomes plus volumineux sur les mêmes IPU.
“L’UIP pourrait devenir le prochain GPU”, a-t-elle déclaré.
L’étude est publiée sur le arXiv serveur de préimpression.
Plus d’information:
Luk Burchard et al, Space Efficient Sequence Alignment for SRAM-Based Computing : X-Drop sur l’IPU Graphcore, arXiv (2023). DOI : 10.48550/arxiv.2304.08662
Informations sur la revue :
arXiv
Fourni par l’Université Cornell
Citation: Un processeur conçu pour l’IA accélère l’assemblage du génome (1er novembre 2023) récupéré le 1er novembre 2023 sur
Ce document est soumis au droit d’auteur. En dehors de toute utilisation équitable à des fins d’étude ou de recherche privée, aucune partie ne peut être reproduite sans autorisation écrite. Le contenu est fourni seulement pour information.