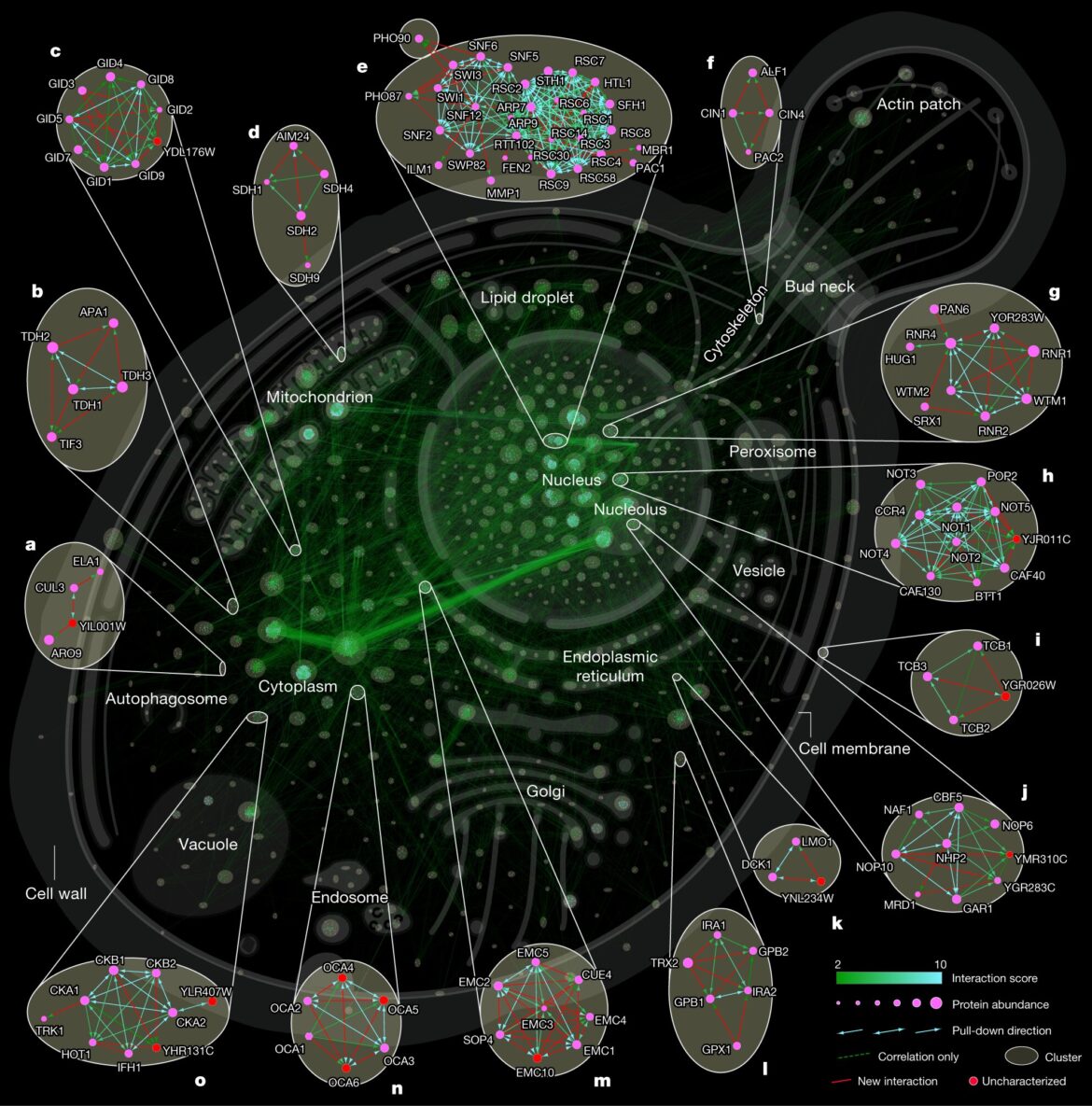
Réseau d’un interactivetome approfondi, mettant en évidence de nouvelles interactions. Carte d’interaction cellulaire de toutes les interactions significatives. Les clusters sont mis en évidence par des cercles et la localisation cellulaire est indiquée par le terme d’ontologie génétique le plus fréquent (composant cellulaire) au sein d’un cluster. a – o, les agrandissements montrent des exemples d’interactions nouvelles ou de celles qui n’ont pas été décrites plus en détail en tant qu’interacteurs potentiels de haute importance et interactions impliquant des protéines non caractérisées. Crédit: Nature (2023). DOI : 10.1038/s41586-023-06739-5
L’expérience révolutionnaire « Six degrés de séparation » de Stanley Milgram a démontré les liens étonnamment étroits qui existaient entre les humains dans les années 1960. Aujourd’hui, l’équipe de recherche dirigée par le professeur Matthias Mann de l’Institut Max Planck (MPI) de biochimie a montré que les protéines de nos cellules sont également bien connectées.
Les résultats de l’étude actuelle, qui sont publiés dans la revue Natureconstituent une avancée décisive dans la recherche sur les protéines et représentent la première vision globale du réseau d’interactions protéine-protéine d’un organisme.
La célèbre expérience de Milgram des années 1960 a montré qu’en moyenne, les gens se connaissent grâce à seulement six relations personnelles. Des analyses plus récentes de réseaux sociaux tels que Facebook ont montré que nous sommes encore plus étroitement liés qu’on ne le pensait initialement, avec une moyenne de seulement quatre « poignées de main » et demie entre nous.
Imaginez chaque protéine de nos cellules comme une sorte de nœud dans un immense réseau social, à l’image des individus dans une société. Tout comme entre les individus d’une société, les connexions et les échanges qu’établissent les protéines sont cruciaux. De plus, ces interactions déterminent, par exemple, si nos protéines peuvent remplir correctement leurs fonctions et tâches et donc si nous restons en bonne santé ou non.
Pour étudier ces interactions et le réseau qui en résulte dans notre corps, les scientifiques ne disposaient jusqu’à présent que de méthodes très incomplètes. Cela rendait presque impossible l’obtention d’analyses approfondies et de résultats significatifs sur le réseau social des protéines.
La méthode du pêcheur
Pour plonger dans ce réseau social de protéines, l’équipe du MPI de Biochimie a utilisé une approche innovante qui permet de filtrer les nœuds individuels du réseau et d’étudier avec quels autres nœuds ils interagissent. La méthode est acronymée AP-MS et combine la spectrométrie de masse avec la purification par affinité, une méthode dans laquelle la protéine cible dont les relations doivent être étudiées est, pour ainsi dire, extraite de la mer de toutes les protéines.
L’affinité pour les protéines avec lesquelles la protéine cible interagit entraîne également leur extraction directe. Le spectromètre de masse peut alors mesurer exactement quelles protéines sont impliquées. De cette manière, ces protéines peuvent être identifiées comme partenaires d’interaction directs de la protéine cible.
Semblable à un pêcheur qui sélectionne l’appât en fonction de sa proie préférée, les chercheurs marquent la protéine cible (appât) avec une petite protéine fluorescente (canne à pêche), qui à son tour est extraite du protéome (mer) par l’anticorps agissant en tant que pêcheur, entraînant avec lui les autres protéines (poissons).
Entrée minimale, sortie maximale
Dans la présente étude, les chercheurs ont réussi à réduire la quantité auparavant importante de 4 litres de matériel cellulaire à seulement 1,5 ml. Comme la croissance des cellules vivantes dans une culture cellulaire prend un certain temps, la nouvelle approche de cette étude permet une étude beaucoup plus rapide et plus précise du réseau protéique dans les cellules. Le nouveau flux de travail offre ainsi des résultats plus approfondis et une représentation cartographiée de l’architecture protéique avec moins d’apport cellulaire et donc moins de temps requis.
L’étude souligne le rôle crucial de la levure en tant qu’organisme modèle en raison de sa comparabilité aux humains. Bien que les levures comptent parmi les formes de vie les plus simples dans la nature, elles partagent avec nous des fonctions cellulaires importantes. Les interactions moléculaires des levures montrent que la grande majorité des protéines sont étroitement liées, chaque protéine formant en moyenne 16 relations, ne les séparant que d’environ quatre « poignées de main ». Les protéines semblent donc agir aussi socialement que nous, les humains, le faisons dans notre réseau social.
Dans la santé et la maladie
“Afin de comprendre exactement où les choses se passent mal dans les cellules malades, il est essentiel de comprendre comment les protéines interagissent entre elles dans les cellules saines. Dans notre étude, nous avons pu découvrir de nombreuses nouvelles connexions entre les protéines également présentes dans les cellules humaines et sont associés à des maladies comme le cancer ou la maladie de Huntington”, explique André Michaelis, premier auteur de l’étude.
Ces recherches sur les partenaires d’interaction de protéines essentielles à notre santé pourraient également servir à tirer des conclusions sur d’éventuelles nouvelles approches thérapeutiques à l’avenir. Cela permettrait d’avoir une approche beaucoup plus individualisée de l’origine des maladies chez chaque personne.
Matthias Mann conclut : « Les résultats de notre étude nous ont donné un aperçu sans précédent du réseau protéique de nos cellules. Je suis convaincu que les connexions et les interactions que nous avons découvertes seront d’une importance cruciale à l’avenir pour le traitement des maladies causées par interactions protéiques défectueuses. Un projet passionnant avec des résultats incroyablement détaillés.
Plus d’information:
André C. Michaelis et al, L’architecture sociale et structurelle de l’interactome protéique de levure, Nature (2023). DOI : 10.1038/s41586-023-06739-5
Fourni par la Société Max Planck
Citation: Une recherche plonge dans le réseau social des protéines (16 novembre 2023) récupéré le 16 novembre 2023 sur
Ce document est soumis au droit d’auteur. En dehors de toute utilisation équitable à des fins d’étude ou de recherche privée, aucune partie ne peut être reproduite sans autorisation écrite. Le contenu est fourni seulement pour information.